リアルタイムPCRのプライマー設計について、実際の画面を使って説明します。
今回、ヒトのp53遺伝子のプライマー作製します。
目的遺伝子のmRNA配列の検索・ダウンロード

① 検索エンジンで「NCBI」と検索し、写真のサイトを開きます。

② 「Nucleotide」を選択します。
③ 「p53 homo」を入力します。
④ 「Search」をクリックします。
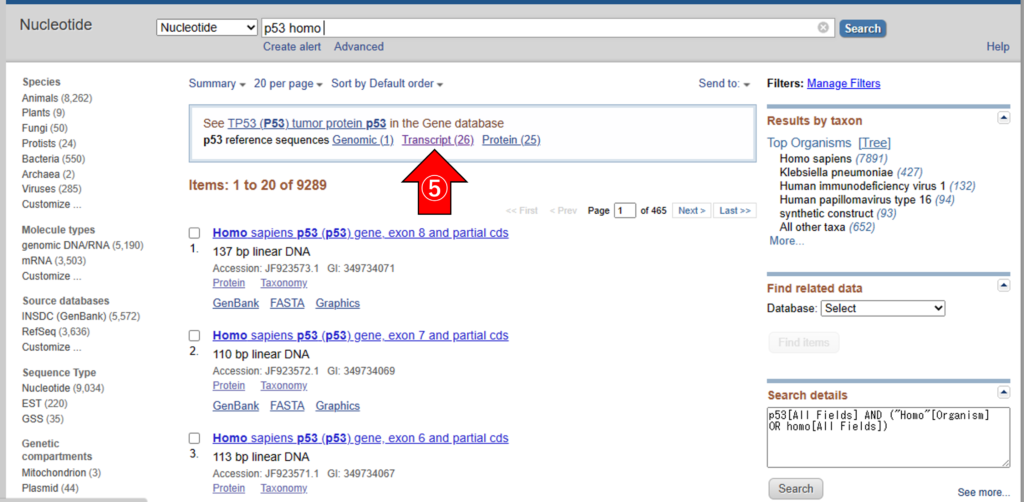
⑤ 「Transcript」をクリックします。

⑥ 「transcript variant 数字」がいくつか検索結果に出てきます。遺伝子ごとに「transcript variant 」の数は異なります。
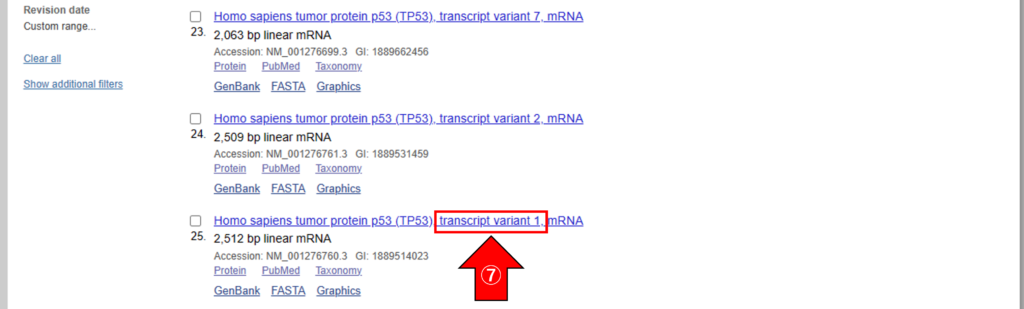
⑦ 「transcript variant 1」をクリックします。
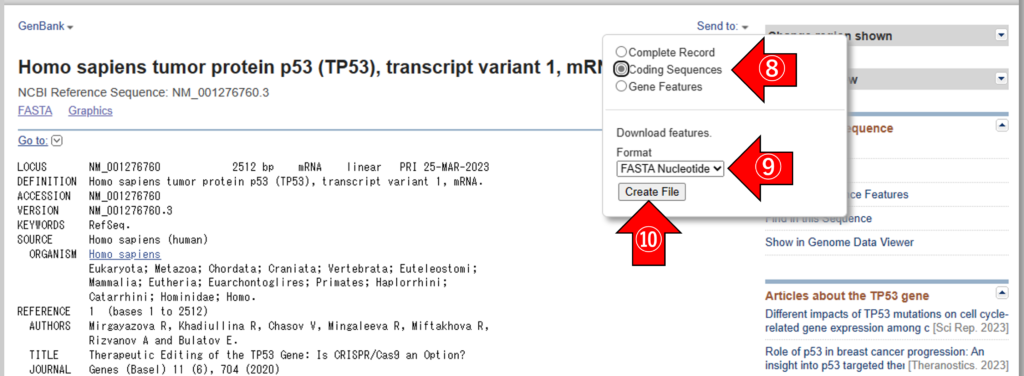
⑧ 「Coding Sequences」を選択します。
⑨ 「FASTA Ncleotide」を選択します。
⑩ 「Create File」をクリックすると、ファイルがダウンロードされます。
プライマーを作製する配列の検索

⑪ ダウンロードしたファイルを開き、塩基配列を選択してコピーします。

⑫ wordを開き、⑪でコピーした配列を貼り付けます。貼り付けた後、改行を消去します。
⑬ ⑥の検索結果全ての「transcript variant 数字」を⑦~⑪の作業を行います。ヒトp53遺伝子の場合、「transcript variant 」が1~13まであるので全て⑦~⑪の作業を行います。今回は分かりやすくするため、「transcript variant 1~4」のみの⑦~⑪を行っています。
⑭「transcript variant 1」の最初の2行をコピーします。
⑮ キーボードの「Ctrl+F」を押し、⑭でコピーした配列を検索します。

⑯ 共通配列に色を付ける等して見やすくします。そして、Variant 1の配列を用いて、順に⑭コピー→⑮検索→目印というように全て行います。このようにして、すべてのVariant内で共通配列が分かります。
*今回、便宜上Variant 1~4までしか行っていませんが、実際にプライマーを作製する場合は、Variant全て行ってください。
Primer3でプライマー作製

⑰ 検索エンジンで「primer3 input」と検索します。
⑱ 「Primer3 Input」をクリックします。
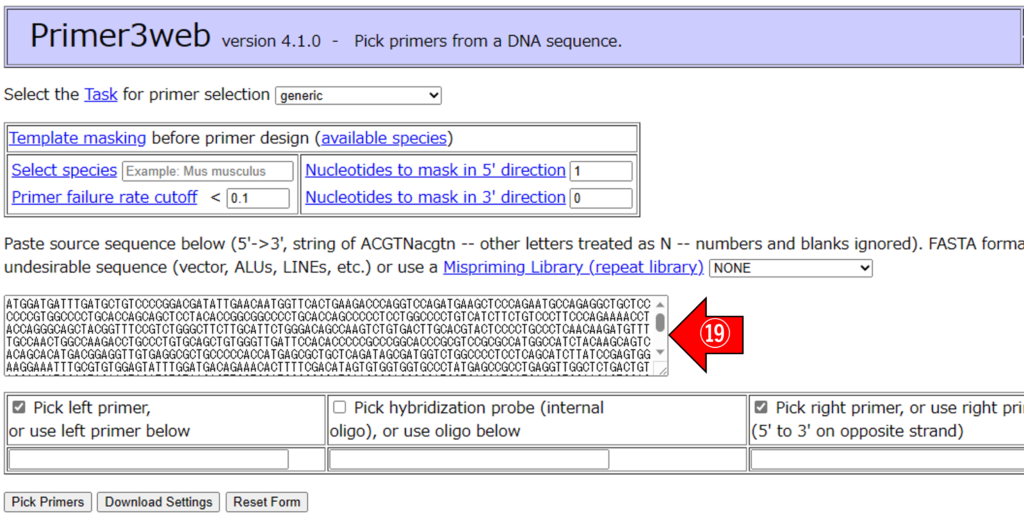
⑲ ⑯で検索したすべてのVariant共通の配列をコピーし、貼り付けます。
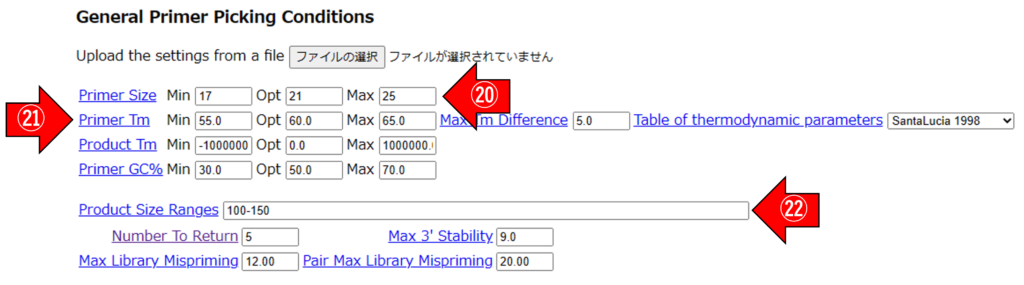
⑳ 「Primer Size」のMin=17, Opt=21, Max=25と入力します。
㉑ 「Primer Tm」のMin=55.0, Opt=60.0, Max=65.0 と入力します。
㉒ 「Product Size Ranges」に100-150と入力します。
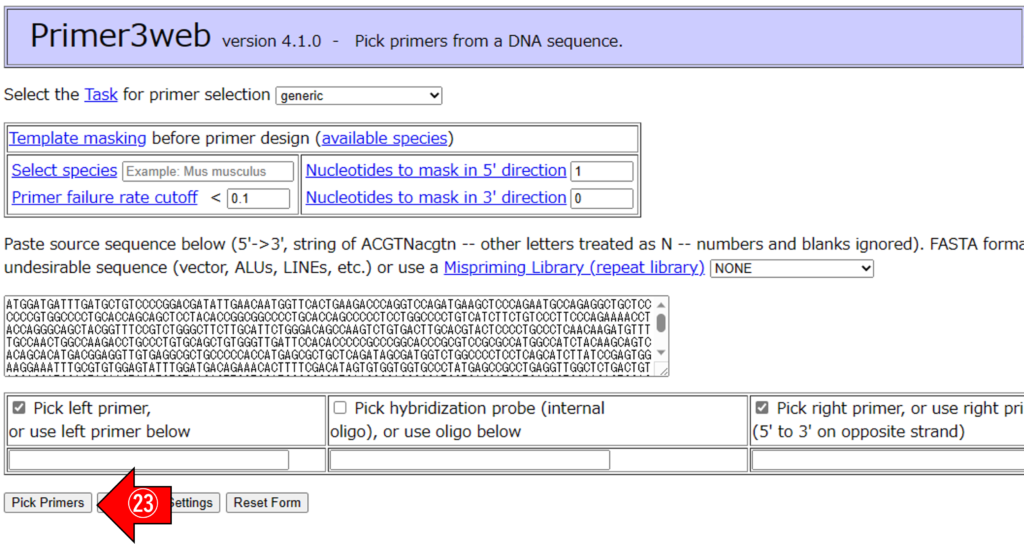
㉓ 「Pick Primers」をクリックします。候補プライマーの結果が以下のように出ます。


UCSC In-Silico PCR
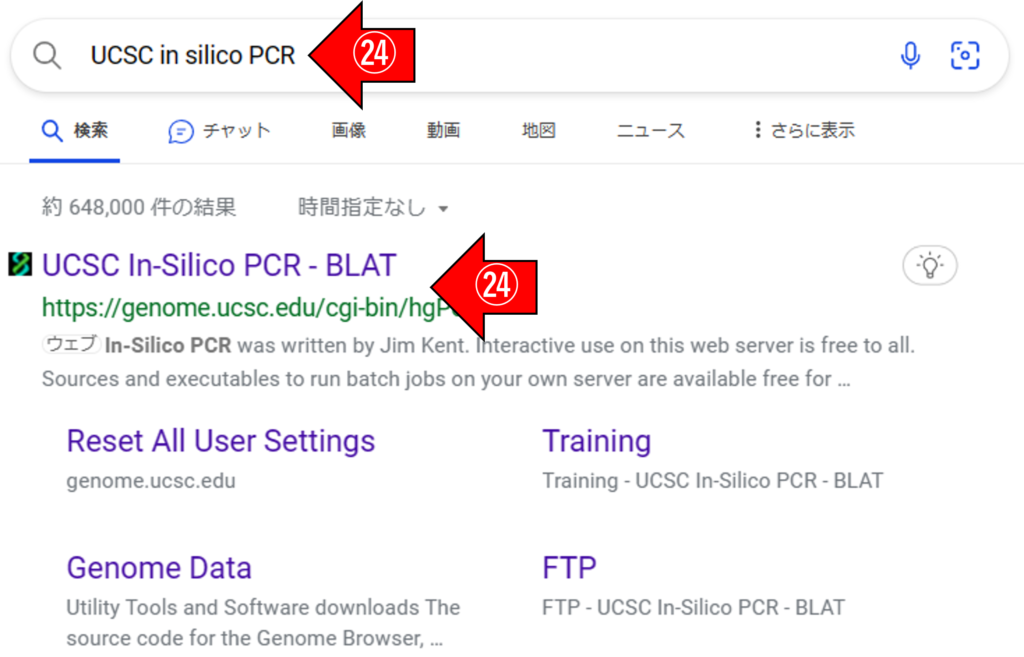
㉔ 検索エンジンに「UCSC in silico PCR」と入力し、結果をクリックします。
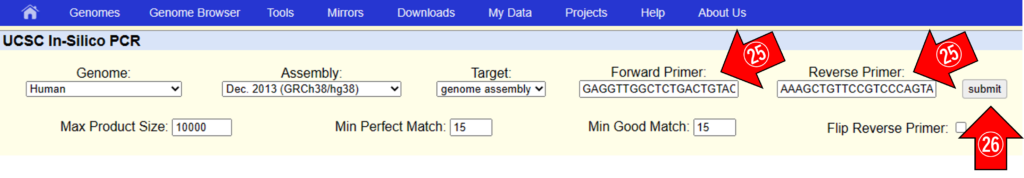
㉕ ㉓の結果の1番最初に出てきたFowerd PrimerとReverse Primerをコピーして貼り付けます。
㉖ 「submit」をクリックします。
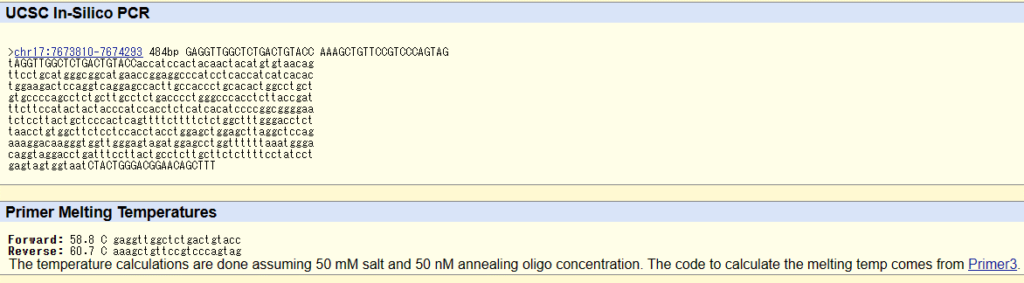
作製したプライマーのProduct sizeが141bpであり、UCSC In-Silico PCRが484bpで異なっているので、イントロンを挟んだプライマーであることが分かります。
もし長さが一致する場合、ゲノムDNAとmRNAが区別できないので、他の候補プライマーをUCSC In-Silico PCRで検索してください。




コメント